5.4 数据标准化 EMP_decostand
在多组学数据分析中,由于不同数据的量纲、量级存在差异,可能导致分析结果出现偏倚。因此,需要采用各种标准化方法来消除这些差异。EMP_decostand继承了vegan包的模块decostand,可以便捷地使用对数比、相对值、中心对数比等方法进行数据标准化。
注意:
模块
模块
EMP_decostand中的参数bySample可以指定按照样本内标准化或者按照特征内标准化。不做特殊设定时,参数bySample将按照vegan包内的建议执行。
5.4.1 微生物数据标准化
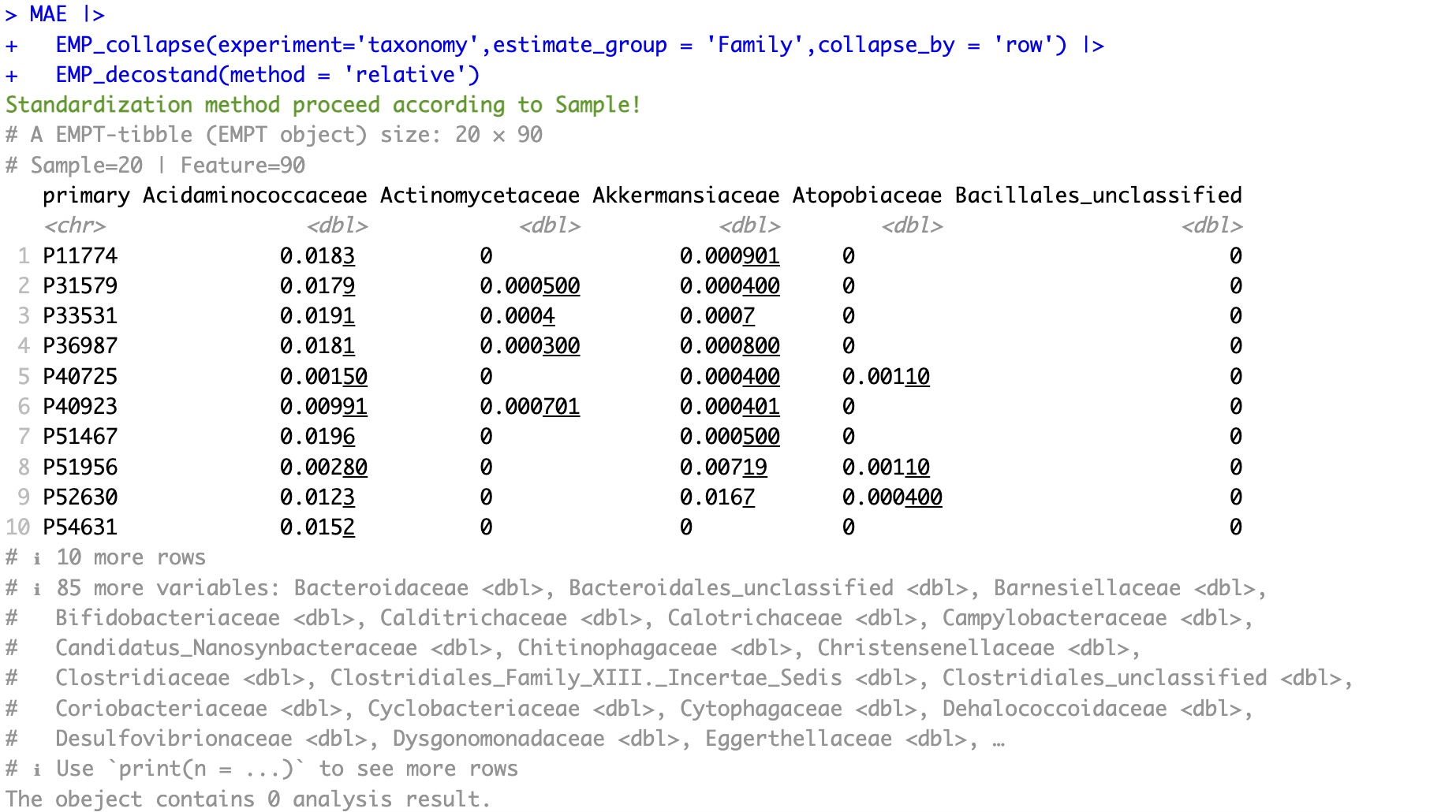
🏷️示例:提取组学项目taxonomy的assay并按照rowdata的Family列进行折叠,将折叠后assay的feature的绝对丰度转化为相对丰度。
MAE |>
EMP_collapse(experiment='taxonomy',estimate_group = 'Family',collapse_by = 'row') |>
EMP_decostand(method = 'relative')
5.4.3 基因组数据标准化
🏷️示例:提取组学项目host_gene的assay并进行log转换。
注意:
①当使用log、pa和integer标准化方法时,无需考虑标准化方向。
②在进行对数转换时,仅对大于0的计数进行处理,0数据则会保持不变。
①当使用log、pa和integer标准化方法时,无需考虑标准化方向。
②在进行对数转换时,仅对大于0的计数进行处理,0数据则会保持不变。
MAE |>
EMP_decostand(experiment = 'host_gene',method = 'log',logbase = 2)

注意:
可以直接使用log2+1的写法,则会对全部数据进行加1,再进行对数转化。也可以省略写成log+1,对数则会根据logbase参数获取。
可以直接使用log2+1的写法,则会对全部数据进行加1,再进行对数转化。也可以省略写成log+1,对数则会根据logbase参数获取。
MAE |>
EMP_decostand(experiment = 'host_gene',method = 'log+1')
